Green Spring Technology: Guaranteeing Your Supply of High-Bioavailability CoQ10 Powder
Coenzima Q10 has broad applications in cardiac health and cosmetic wellness, yet its practical efficacy remains constrained by two core challenges: low bioavailability and inconsistent supply quality. Traditional extraction methods yield low yields, while chemically synthesized products exhibit insufficient activity, severely limiting the performance of end products and market trust.
Green Spring Technology systematically addresses these pain points through a tripartite technical system combining:
✨ High Bioavailability
Optimized microbial strains and metabolic pathways through synthetic biology yield a fully trans-isomeric active structure easily recognized by the human body, ensuring absorption efficiency from the source.
✨ Stable, Scalable Supply
Standardized fermentation processes and parameter control guarantee consistent quality across batches, supporting long-term product development and market planning for clients.
✨ Full-chain traceability
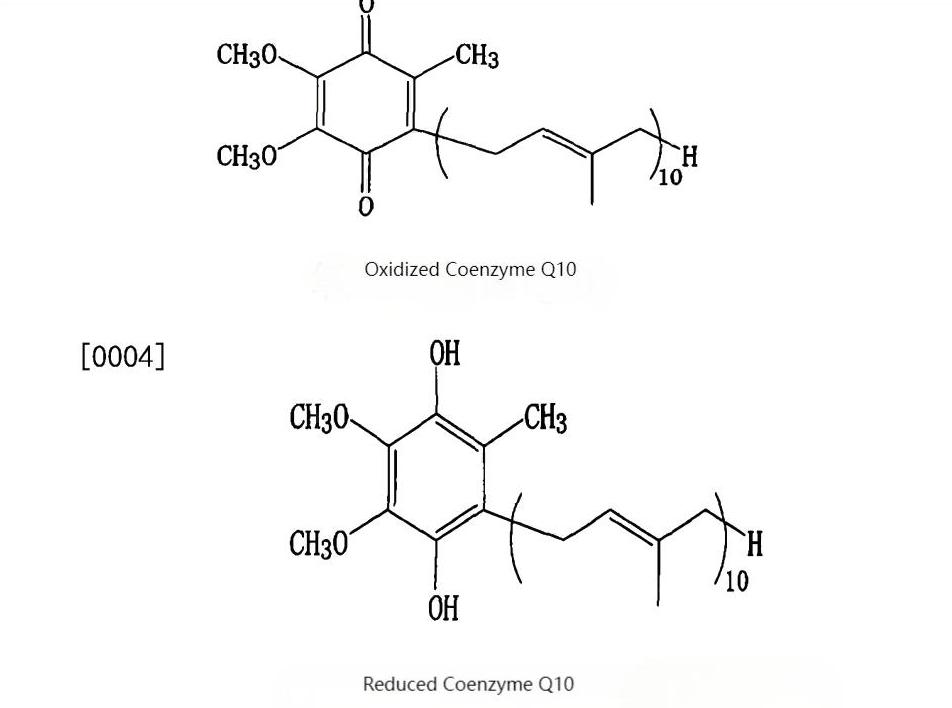
Establishes critical data records from microbial strains to finished products, providing clients with transparent, credible raw material quality profiles to support product registration and market communication.
Green Spring Technology offers pharmaceuticals, health foods, and premium skincare brands:
★ High-potency, easily absorbable CoQ10 raw materials
★ Sustained, stable, large-scale supply assurance
★ Comprehensive, verifiable quality traceability system
Green Spring Technology: Achieving stable supply of high-potency CoQ10 through precision biosynthesis and fermentation technology
Leveraging synthetic biology and precision fermentation, Green Spring Technology systematically builds an end-to-end technical framework from molecular design to industrialization, dedicated to delivering CoQ10 raw materials with high bioavailability and consistent quality.

1 Precise Biosynthetic Pathway Regulation
1.1 Aromatic Ring Synthesis Pathway Control
· Utilizes genetically engineered strains to optimize the conversion pathway from branched acids to polyhydroxybutyrate (PHB)
· Regulates ubiA/coq2 gene expression to enhance PHB-polyisoprenyl transferase activity
· Precisely control hydroxylation, methylation, and decarboxylation reaction sequences to ensure correct quinone ring modification
1.2 Specific Synthesis of Polyisoprene Side Chains
· Select strains with specific side chain synthesis capabilities to precisely control polyisoprene side chain length to 10 units
· Optimize the MEP metabolic pathway to enhance IPP/DMAPP precursor supply efficiency
· Regulate isoprenyl pyrophosphate synthase expression to achieve C50 side chain specificity
2 Green Spring Technology Coenzyme Q10 Raw Material Precision Fermentation Process System
2.1 Professional Strain Breeding
· Optimize Gram-negative bacteria as base production strains based on microbial CoQ10 distribution patterns
· Employed directed mutagenesis and rational screening strategies to obtain high-yield mutant strains
· Engineered strains achieved industry-leading fermentation yields under optimized conditions
2.2 Medium System Optimization
· Precise carbon source ratio: Utilized C10-C13 alkanes as optimal carbon sources
· Optimized nitrogen source selection: Determined peptone as the most suitable nitrogen source (16 g/L)
· Precise metal ion regulation:
Mg²⁺ (12.2 mmol/L) enhances enzyme activity
Fe²⁺ (1.8 mmol/L) boosts electron transfer
Mn²⁺ (0.9 mmol/L) optimizes metabolic pathways
· Targeted precursor supplementation: propionic acid, isoamyl alcohol, geraniol, p-hydroxybenzoic acid
· Application of natural effectors: Natural substances rich in synthetic precursors, such as soybean oil, soybean meal, and carrot juice
2.3 Precision Control of Cultivation Conditions
· Light regulation: Established light-anaerobic cultivation mode, increasing yield by over 60%
· Precise dissolved oxygen control: Implemented customized DO strategies for different microbial strains
· Standardized inoculum volume: Optimized inoculum at 4% to shorten fermentation lag phase
· Precise pH regulation: Maintained fermentation pH at 5.0 to maximize intracellular accumulation
2.4 Process Control and Quality System
· Online monitoring system for real-time regulation of critical parameters (temperature, pH, DO)
· Dynamic optimization of synthetic pathway flux via metabolic flux analysis
· Standardized operating procedures to ensure batch-to-batch quality consistency
· Verify product purity and structural integrity via HPLC and other analytical methods
· Implement end-to-end quality monitoring with full traceability from strain to finished product
3 Green Spring Technology's Coenzyme Q10 Technical Advantages and Core Value
3.1 Product Characteristic Advantages
· High Bioavailability: All-trans active structure identical to human endogenous CoQ10
· Stable Supply Assurance: Precision fermentation control ensures high batch-to-batch quality consistency
· Complete Traceability: Quality documentation system established from gene to finished product
3.2 Industrialization Assurance
· Stable Yield: Multi-parameter synergistic optimization ensures production stability
· Activity Assurance: Optimized cultivation conditions maintain high product bioactivity
· Efficiency Enhancement: Standardized processes significantly improve production efficiency
· Cost Optimization: Scientifically formulated to effectively control production expenses
Through deep integration of synthetic pathway regulation and fermentation process optimization, Green Spring Technology has established a comprehensive industrialization technology system. We provide pharmaceutical, health supplement, and cosmetics industry clients with premium, reliable CoQ10 raw material solutions, empowering partners to enhance product competitiveness and jointly drive innovation in the wellness industry.
4 Green Spring Technology Drives New Breakthroughs in CoQ10 Raw Materials via Synthetic Biology and Precision Fermentation
As a key substance in cellular energy metabolism, CoQ10 sees continuously growing demand across pharmaceuticals, health supplements, and cosmetics. However, traditional production methods face industrial bottlenecks such as limited fermentation capacity and complex extraction processes.
Through dual breakthroughs in precise synthetic pathway regulation and systematic fermentation process optimization, Green Spring Technology has successfully achieved stable, large-scale production of high bioavailability CoQ10. We have not only established a complete technological system from genetic modification to fermentation scale-up, but also significantly enhanced product yield and quality stability through innovative extraction and purification processes.
Moving forward, we will continue to deepen our technological focus in the following areas:
1. Synthetic Biology Innovation: Further optimize metabolic networks through multi-gene synergistic regulation
2. Intelligent Fermentation Control: Achieve real-time, precise regulation of fermentation parameters using process analytics
3. Green separation processes: Developing efficient, eco-friendly extraction and purification routes to enhance product competitiveness
Green Spring Technology is committed to providing global customers with stable, high-bioavailability CoQ10 raw materials, driving high-quality development in the wellness industry. Contact us at helen@greenspringbio.com or WhatsApp: +86 13649243917 to obtain samples and technical documentation, and together we will pioneer new applications for CoQ10.
Referencias:
[1]Littarru G p . Biomedical and clinical aspects of coenzyme Q [J]. Clin Investig, 1993, 71(8):587-588.
[2] Yuan Jing, Wei Hong. Progress of microbial fermentation for coenzyme Q10 production[J]. Amino Acids and Bioresources, 2004, 26(1):53-56.
[3] Wu Zufang, Weng Peifang, Chen Jian. Progress in the study of the function of coenzyme Q10[J]. Journal of Ningbo University: Science and Technology, 2001, 14(2):85-88.
[4]Wang Genhua, Qian He. Coenzyme Q10 and its health benefits[J]. Jiangsu Food and Fermentation, 2002,(2):16-17.
[5]Huang W, Xu JZ. New Progress of Coenzyme Q10 Research[J]. Henan Chemical Industry, 2003, 223(2): 12-15 .
[6]Maktoto K . Biosynthesis, bioproduction and novel roles of ubiQuinon ae [J].J Biosci Bioeng, 2002, 94(6):511-517 .
[7]Meganathan R. ubiQuinone biosynthesis in microorganisms[J]. ubiQuinone biosynthesis in microorganisms [J]. FEMS Lett, 2001, 203(2):131- 139 .
[8]poon W W, Davis D E, Ha H T, et al . Identificación de Escherichia coli ubi B, un gen requerido para el primer paso de la monooxigenasa en la biosíntesis de ubiquinona [J]. J Bacteriol, 2000, 182(18):5139-5146.
[9] Rohmer M . El descubrimiento de una vía independiente del mevalonato para la biosíntesis ISO - prenoide en bacterias, algas y plantas superiores [J]. Nat prod Rep, 1999, 16(5):565-574.
-
anterior
The Power of Fermented CoQ10 Ingredient: Empowering Advanced Health Supplements
-
siguiente
Green Spring Technology Supplies Yeast-Derived Glutathione Ingredient Solutions


 inglés
inglés francés
francés español
español ruso
ruso coreano
coreano Japonés japonés
Japonés japonés



